【摘要】 lammps官方也推荐的一个开源建模软件———Moltemplate。
上期我们已经列举了模拟计算中分子建模的4种方式,今天我们再补充一种lammps官方也推荐的一个开源建模软件———Moltemplate。
为了不因笔者的翻译造成误解,这里把完整的的软件官方描述贴上:
LAMMPS is an extremely flexible and customizable molecular dynamics engine. Moltemplate is a general cross-platform text-based molecule builder for LAMMPS. Moltemplate was intended for building custom coarse-grained molecular models, but it can be used to prepare realistic all-atom simulations as well。
从上面的描述中我们可知:Moltemplate是专门为lammps建模而开发的一款开源软件,既可以建立粗粒化模型,也可以建立全原子模型。Moltemplate软件官方有一个专门的Manual(http://www.moltemplate.org/页面的Docs),内容很多,今天我们用1个例子来简单介绍Moltemplate的用法。(Moltemplate的安装说明见软件内README文件)
以Water mixed with sodium & chloride ions为例
需要按照Moltemplate语法准备3个文件spce.lt、ions.lt、system.lt
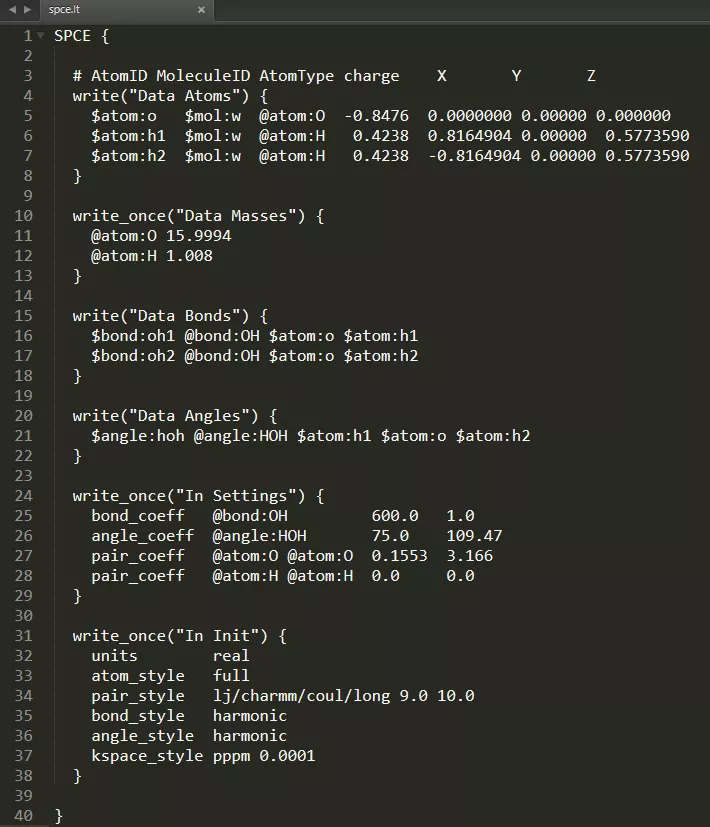
1.spce.lt:
spce.lt:SPEC {}定义了SPCE水分子,{}内为具体的分子类型、原子类型、原子电荷、原子坐标(4-8行);原子质量(10-13行);键类型(15-18行);键角类型(20-22行);力场与一些通用设置(24-38行)。细心的小伙伴已经发现,lt文件内容与lammps的data文件需要的内容一致,是的!的确是这样。
.webp)
2.ions.lt
类似的,ions.lt:NaIon{}与ClIon{}定义了2个分子。
.webp)
3.system.lt
system.lt:1-2行导入了之前的两个lt文件;4-6行以SPCE水分子为模板创建了1000个SPCE水分子并进行了平移操作;8-10行以NaIon分子为模板创建了8个NaIon并进行了平移操作;12-14行以ClIon分子为模板创建了8个ClIon并进行了平移操作;16-17行对所有NaIon、ClIon分子进行了附加的平移操作防止与水分子重叠;19-23行定义了x、y、z三维方向上的范围,即确定模拟体系的空间范围。
以上3个lt文件都准备完毕后,运行moltemplate.sh system.lt即可生成data文件(moltemplate.sh需加入环境变量)。下图为运行后文件夹内容。
.webp)
今天只是以1个例子来简单介绍Moltemplate的用法,事实上Moltemplate软件可以生成非常复杂的结构,比如下图的蛋白质,但需要花一定的时间和精力深入学习Moltemplate。
.webp)
本文所有内容文字、图片和音视频资料,版权均属科学指南针网站所有,任何媒体、网站或个人未经本网协议授权不得以链接、转贴、截图等任何方式转载。











 您已经拒绝加入团体
您已经拒绝加入团体

 2021-08-02
2021-08-02
 9288
9288
 0
0










